只有当研究人员们确定了一个基因都由哪些东西组成的时候,他们才有可能计算出人类拥有多少基因。与此同时,各种各样的基因狩猎程序正在变得越来越复杂和精致——
几十年来,研究人员们一直在计数人类的基因数量,但这些计算结果还都不理想。几年前,人类基因数量的最佳估计值高达10万个,当人类基因组序列发表时,这个数字降到了3万个,最近又跌到了2万。研究人员们称,若要完全发掘和发挥人类及其他基因组测序结果的效用,他们需要一个更好的人类基因计数值。
3年前,基因组测序界圈内开始了一项称为“Gene Sweep"的赌注式台球游戏,对一且人类基因测序完成后可能产生的人类基因数量进行打赌。今年夏,研究人员们承认他们还远远没有接近获得最后的计数值。他们决定中止这项悬案,认为这个计数值平衡在24500个蛋白质编码基因左右。
在美国加州圣克拉拉的Affymetrix公司的一位计算机科学家大卫 · 库尔普(David Kulp)认为:问题关键在于如何定义一个基因,“很难确定地讲什么是正确的、什么是错误的。”分子生物学家们正在发现,一些基因的长度比任何人所预期的长度还要短。有时候,很难确定一段基因密码是一个独立的基因还是两个重叠的基因。对于如何区分编码多个蛋白质的基因和只编码RNA的基因样序列,学术界内还不大能确定。
这种复杂性已经使生物信息学家们认识到他们编写软件的能力的局限性。在这个仅有10年历史的领域内,人们设计了计算机程序来分析DNA序列数据,其中还包括检测出有关基因。它们的数学内涵越来越复杂而精细,它们的算法还充分考虑和收集了遗传学家们最好最新的关于各种基因和各种蛋白质的知识,以及分子生物学家们对各种基因是如何隐藏在DNA中的认识。这些计算机专家中,有些还甚至开始进行他们自己的实验,以便更好地识别各自的有关基因。
他们有许多工作要做。通常,他们可以识别出编码一个蛋白质序列的一段DNA,但是基因的大小、数目以及蛋白质编码和非编码区域在该基因中的精确分布仍然难以分辨。这些情况中最令人困惑的是所谓的“暗物质”(dark matter),即在基因组中似乎是无基因但可能包含隐藏的编码序列的区域。
令人困惑的成果
20世纪30年代,乔治 · 比德勒(George Beadle)和爱德华 · 塔吐姆(Edward Tatum)提出:一个基因只编码一个蛋白质,这种假说在其后几十年间一直是人们的——种常识。现在,人们已经知道这种假说过于简单化了。一个基因既可以产生多种蛋白质分子,也可以只转录成为RNA分子而不是蛋白质分子。
人类基因中的蛋白质编码区被称为外显子,它们仅占DNA总量的2%,在其余98%中则不存在外显子。有些奇怪的遗传性质也使基因计数复杂化。一些非常简单的基因只由一个外显子组成,它们非常微小,极易于被人工和计算机基因计数器(软件)所忽略。相反地,那些由于其DNA组成发生某些畸变而不再产生功能的、所谓的“假基因”......则人为地夸大了基因的数量。
在目前估计的24500种基因中,“有3000个可能是假基因,”设在英国剑桥大学的欧洲生物信息学研究所的一位首席基因统计专家伊万 · 伯尔尼(Ewan Bimey)如是说。很多人在坚持研究、试图确定哪些基因是真基因,伯尔尼就是其中之一。华盛顿大学圣路易斯分校的一位计算机科学家米歇尔 · 布伦特(Michael Brent)说:“我相信所有的基因预测程序都受此影响,如果我们考虑了假基因的因素在内,每个人或软件的预测值都会大大得到改善。”
更糟糕的是,已经证实:对于基因组的某些部分,即使是最好的基因预测程序也完全不能测出,它们包括暗物质,这些区域由于“明显地毫无基因”而得名。西班牙巴塞罗那市的Pompeu Fabra大学的一位计算生物学家罗德里克 · 盖戈(Roderic Guigo)说。基因预测程序对这些暗物质一无所知,许多人担忧这种空白包含了许多研究者所看不到的基因;盖戈还指出:“暗物质基因可能拥有与我们所能识别的基因不同的特征。”
一个基因......两个基因......
任何具有实用价值的基因测序都必须在发现暗物质区域以外的基因方面切实有效。一般说来它们通过两种方法之一来达此功能:。“从头开始”(abinitio)方法通过检测到DNA序列中的特殊的模式(Pattern)来识别基因,这些模式诸如那些标示各种基因的起点和终点的模式。另一种方法是比较方法:它们根据它们与已知蛋白质和基因的相似性来发现新基因。这两种方法产生了一种金发人(Goldilocks)两难悖论:“从头开始”型软件把任何看起来模模糊糊地像一个基因的东西都归类为一个基因,所以它们对基因总数的估计是偏高的;比较法不能识别那些不相似的基因,故它们的估计值是偏低的。似乎还没有什么方法可以不多不少地估计出基因的数目来。华盛顿大学圣路易斯分校的一位计算机科学家加里 · 斯托摩(Cary Stormo)则认为:“但是各种方法都比它们过去有了巨大的进步。”
许多人把这一领域的开始回溯到一种名为“基因模建器”(Gene Modeler)的“从头开始”型软件,它是1990年由当时在拉斯克鲁西斯的新墨西哥州立大学的两位科学家:克里斯菲尔德斯(Chis Fields)和卡里 · 索德兰(Cari Soderlund)在寻找美丽隐杆线虫基因时编制的。那时存在的其它软件比较笨拙,较少采用直接的方法。例如,BLAST和FASTA两种程序要把DNA序列翻译成蛋白质序列,再与已有的蛋白质数据相比较。
这个领域发展很快,早期的基因预测者还包括盖戈,他在Gene Modeler方法的基础上建立了GeneID来寻找人类基因(而不是蠕虫基因)。1991年,斯托摩的一位研究生埃里克施奈德(Eric Snyder)编写了一套名为Gene Parser的软件,它吸收采用了一种称为必威在线网站首页网址 编程(dynamic programming)的技术,从内含子、即从不编码蛋白质的基因区域中分离外显子。
令人遗憾的是,施奈德没有将他的工作进行到底。马里兰州罗克威尔的基因组研究所(TICR)的斯蒂文 · 萨尔兹伯格(Steven Salzberg)及其同事最近出品了几个新的程序,其中之一合并了非常多的背景信息以产生预测结果,例如关于外显子和内含子大小的清洁器原则(clearer rules)。另一种程序可以同时针对两个完整的基因组,通过计算可以对两个基因组进行对比并找到双方的匹配起始点。
许多研究者现在采用这后一种方法,因为相似的物种倾向于拥有非常相似的序列的基因。这些相似基因的蛋白质编码区域往往相互匹配,并从而从包围它们周围的、不匹配的无意义序列中凸现出来。
一种名为GENSCAN的软件从其他软件中脱颖而出,成为该领域的标准。当麻省理工学院(MT)的克里斯 · 伯格(Chis Burge)在1996年开始编写该软件的时候,他的许多同行都主张采用比较法。他们通过将DNA与已有数据库中的已知基因进行匹配,来在新测序的基因组中找到基因。但是伯格不同意这种方法,他提醒说:“我们拥有的序列是人类序列,实在是没有什么可以与之相比较的。”到那时为止,还没有其他的脊椎动物基因(被测序),而那时果蝇、线虫和其他微生物的已测序基因组中的匹配区域还是相当有限的。
与这些同行不同的是,伯格从加州大学克鲁兹分校的一位计算机科学家大卫 · 豪斯勒(David Haussler)那里吸取了一条经验。在那时的三年前,豪斯勒就认为基因预测问题是基因组学界所面临的重要挑战,就如同语言学家们试图找出句法、语法和语言的其他特征的某些固定模式那样。他和其他一些人建议他们的同事从语言学中引人——种统计学工具,称为隐匿型马尔科夫模型(Hidden Markov Model)。该模型使软件在从已有信息中得到的一系列基准问题的基础上进行预测。
伯格指出:“现在,我们已有了一系列区分各种基因的各个部分的模式和规则。”例如,所有的,或者至少是绝大多数基因都有特征性的起始和终止序列。在外显子的末端还拥有一段特征性的序列,它可以告诉一些酶切断后续的内含子序列。伯格“使”这些模型“学会”这些规则,并用此模型分析了几百个已知内含子和外显子区域的基因的序列。这些分析所得的模式成为了隐匿型马尔科夫模型的一个典范,它可以预测一段DNA分子中是否包含基因。
经证实,该方法取得了巨大成功。现在,隐匿型马尔科夫模型已成为了大多数基因预测算法的标准。就GENSCAN本身而言:“它比那些没采用隐匿型马尔科夫模型的软件表现出巨大的优越性。”萨尔兹伯格说。而布伦特也说:“它是最棒的。”
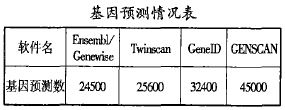
但是,GENSCAN即使是最好的,也有它的缺点,它在基因识别中显得过于灵敏:它预测人类基因组有4.5万个基因,几乎是现在为多数人所接受的总数的两倍。伯格承认GENSCAN存在其自身的问题,但他认为基因过多要比基因过少好,因为人们总是可以排除掉假阳性的。
伯格说:GENSCAN很可能永远也不能预测出正确|的基因数目。自从该软件引入后,基因组学界发生了巨大变化。对于得到正确的基因计数来讲,将来自人类和其他物种的新的序列数据考虑在内,可能是至关重要的。“如果我现在做的工作是基因发现,我将选择的是比较法。”他讲。

人类基因究竟有多少?
包括官方的GeneSweep counter软件、Ensembl / Genewise软件在内的几种软件,都根据与已知基因的相似性来发现基因。但是它们比早期的比较法软件获得了高度的发展。伯尔尼和他的同事们开发的Genewise软件在已知蛋白质的基础上工作。这些蛋白质来自那些其成员的氨基酸序列及其DNA序列或多或少相似的蛋白质家族。利用这些家族相似性,Genewise软件就可以将新的蛋白质序列与那些以前发现的来自相同或不同生物物种的蛋白质及其基因进行比较了。
由于被测序的基因组种类越来越多,人们越来越容易地对整个基因组进行匹配而不只是比较序列中的短片断,所获成果也越来越多。Affymetrix公司的库尔普说:这些比较方法是目前最有希望的技术路线。一些编程小组正在将多种基因预测策略合并起来。
尽管有了这些进展,下一代程序中也很难有得到相近的基因总数。“暗物质”若隐若现、仍是一个巨大的谜,目前尚无程序可揭开它的面纱。为了揭示它,我们需要了解为什么各种基因有其各种特征,以及各种“暗物质”基因会是什么样子。简而言之,盖戈说:“为了改善我们的软件,我们需要更好地认识各种基因的生物学知识。”没有人敢预言这方面会发生什么。
[Science,2003年8月22日]